De novo Bacterial Sequencing
特長
・バクテリアを対象とした全ゲノムシーケンシング解析。
・経験豊富なCJ Bioscience社の微生物ゲノム受託解析サービスをご提供。
・ゲノム配列の比較解析サービスもご提供可能。
・解析データは、CJ Bioscience社独自のウェブツールを使って自由に閲覧、論文や研究発表用に加工することが可能。
解析概要
ゲノム配列未知のバクテリアを対象として、全ゲノムの塩基配列を決定します。複数機種の次世代シーケンサー(NGS)をご用意していますので、研究目的に適したシーケンシング方法が選択可能です。ゲノムの機能解析、進化、比較ゲノムや薬品開発・エネルギー産業研究に応用していただけます。

解析結果
解析データは、オリジナルウェブツールEzBioCloud Whole Genome Analysis (WG)※1およびComparative Genomics (CG)※1, 2を利用して、下記の内容を閲覧・分析していただけます。
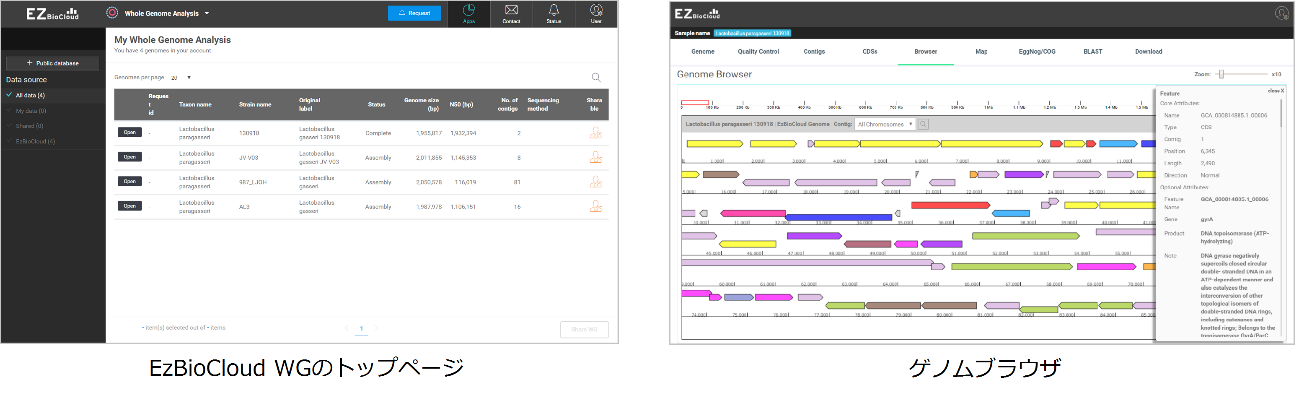
■ Whole Genome Analysis:サンプルごとの全ゲノムシーケンシング解析
・de novoアセンブリングによるコンティグ作成
・予測されたCDSへのアノテーション付与
・予測されたCDSのゲノムブラウザおよびゲノムマップ表示
・BLASTを用いたホモログ探索
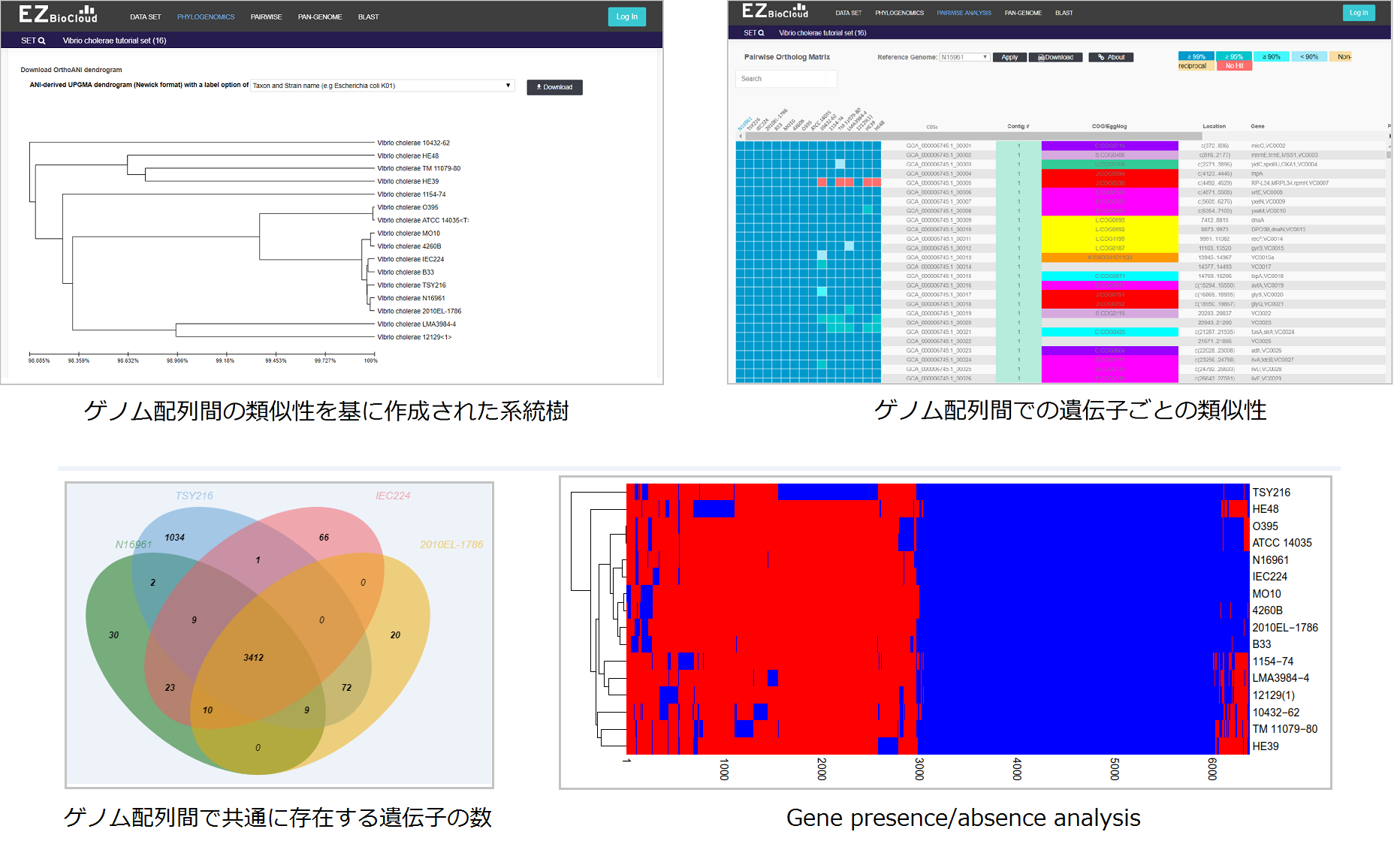
■ Comparative Genomics:お客様ご提供のゲノム配列及び公共データベース上のゲノム配列を用いた比較解析(オプション)
・ゲノム配列間の類似性を基に作成された系統樹
・リファレンスと任意のゲノム配列間の遺伝子ごとの類似性
・複数ゲノム配列間で共通に存在する遺伝子の数
・遺伝子構成によるクラスタリング解析とヒートマップ作成(Gene presence/absence analysis) など
※1 ご使用になる際は、WebブラウザとしてGoogle Chromeの使用を推奨します。
※2 詳細はCJ Bioscience社ホームページのサイトをご参照ください。
納品物
・解析結果(EzBioCloud WGおよびCGを使用して納品後1年間、閲覧または各種ファイル取得可能)
・リード情報(FASTQ、EzBioCloudデータダウンロードサイトより納品後1年間取得可能)
・品質評価結果
サンプル条件
| 機種 | MiSeq | PacBio |
|---|---|---|
| サンプルの種類 | 精製ゲノムDNA | 精製ゲノムDNA※1 |
| DNA量 | 1 µg | 20 µg |
| DNA濃度※2 | 50 ng/µL以上 | 150 ng/µL以上 |
| 溶液量 | 20 µL以上 | 20 µL以上 |
| OD260/280 | 1.8以上 | 1.8以上 |
| 溶媒※3 | 10 mM Tris-HCl (pH 8.5) またはNuclease-free水 |
10 mM Tris-HCl (pH 8.5) またはNuclease-free水 |
※2 サンプルの定量はQubitまたはPicoGreenを用いた方法を推奨しております。
※3 EDTAを含まないバッファーを溶媒としてご使用ください。
解析例
| 解析内容 | 5 Mb以下ゲノムのドラフトシーケンシング |
|---|---|
| 機種 | Miseq |
| リード長 | 300 bp |
| シーケンス方法 | Paired End |
| バイオインフォマティクス解析 | アセンブリングによるコンティグの作成、コンティグ上のORF予測、 予測されたORFへのアノテーションなど |

