「Riken genesis Visualizer for Proteome」の活用

みなさん、こんにちは。理研ジェネシス営業部です。
Olink Visualizerでデータを可視化
理研ジェネシスのOlinkプロテオーム解析では、血清・血漿、脳脊髄液、細胞外小胞(EVライセート)などを対象に、Olink技術を活用して高精度なタンパク質発現解析を提供しています。Olinkの発現変動データを用いた統計解析を通じて、新規バイオマーカーの発見や、生体内で起きている変化を深く捉えることができます。
弊社ではOlinkのデータ取得だけではなく、バイオインフォマティクス解析のサービスもご用意しています。Olinkのデータを用いて、群間のタンパク質発現比較、発現変動が上位のタンパク質の抽出、クラスタリング、パスウェイ解析、データの可視化(散布図、クラスタリングなど)などを実施します。さらに、これらの解析データは、弊社独自のOlink Visualizerで納品します。
Olink Visualizerではユーザーフレンドリーに統計解析データを可視化し確認することができます。下図はOlink Visualizerの一部をスクリーンショットしたものです。2群間比較の数値データ(上段)、Volcano plot(中段左)、各タンパク質の発現相対値のBar plot(中段右)、クラスタリング結果のヒートマップ(下段)を示しています。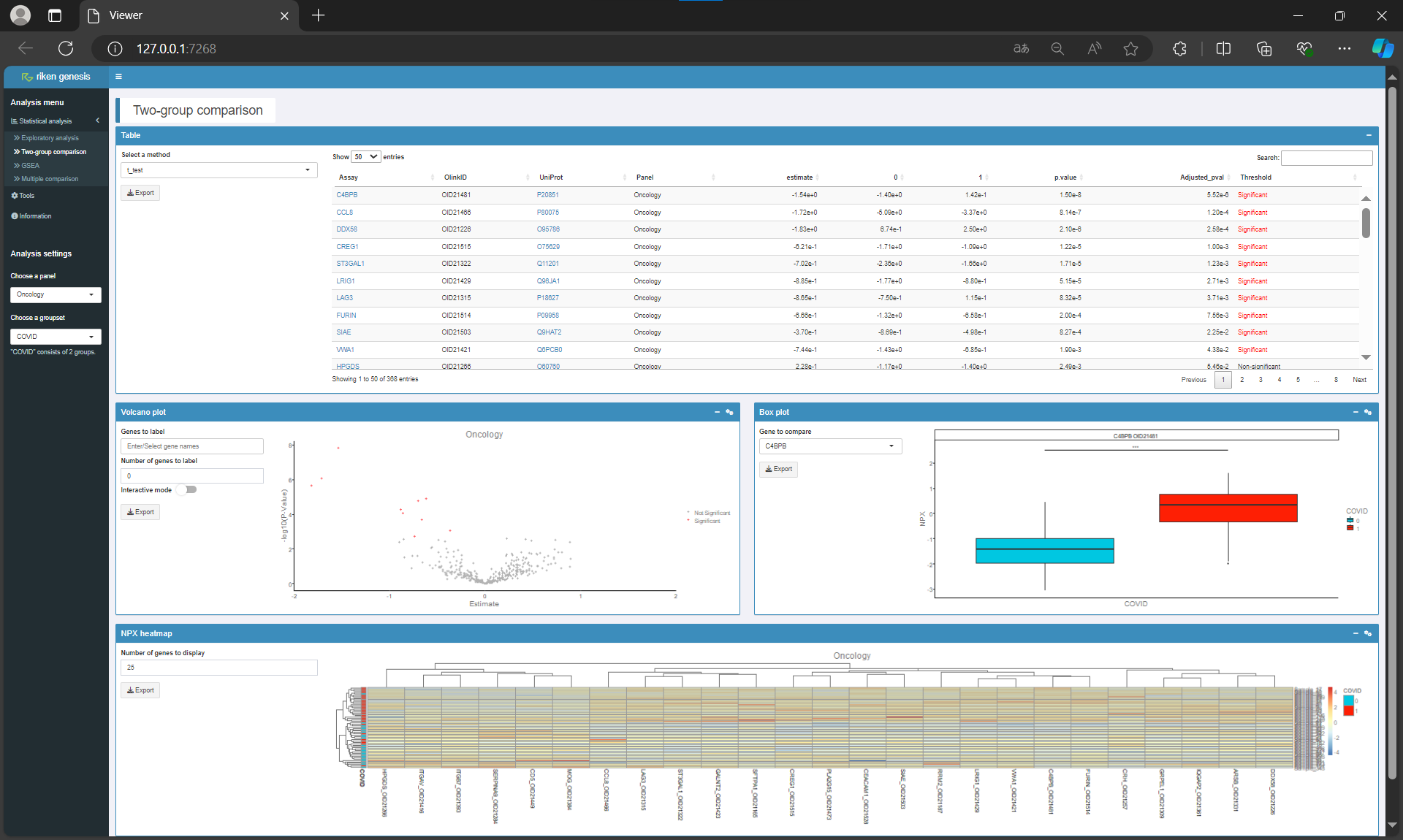
Olink Visualizerによるデータ可視化の実践ガイド
弊社HPでは、お客様のお悩みや課題に合わせて、Olink Visualizerを用いたデータ可視化のポイントをわかりやすく解説した3~5分程度の動画をご用意しています。
各動画の内容を簡単にまとめましたので、ご覧ください。→ 動画・資料一覧(遺伝子解析受託)
① 全体的な発現プロファイルが群間で異なるか調べたい
解析方法:PCA(主成分分析)・UMAP
数千ものタンパク質発現プロファイルを低次元に要約し、サンプル間の差異や類似性を可視化することができる解析です。
② 二群間で発現量が異なるタンパク質を調べたい
解析方法:t検定による二群間比較解析
補正後のP値を指標に、統計的に優位な発現変動を示したタンパク質を同定することができます。
③ 発現変動の生物学的な意義を調べたい
解析方法:Gene Set Enrichment Analysis (GSEA)
GSEAは発現解析の研究で広く使用されている手法で、t検定による二群間比較解析の結果を用いた解析です。タンパク質の発現量変化と関連のあるパスウェイなどを同定することができます。
④ 多群間で発現量が異なるタンパク質を調べたい
解析方法:ANOVAによる多群間比較解析
分散分析(ANOVA:Analysis of Variance)は、3つ以上の群で比較解析を行う場合に用いる手法です。補正後のP値を指標に、統計的に優位な発現変動を示したタンパク質を同定することができます。
このように理研ジェネシスのOlinkデータ解析オプションサービスは一般的な統計解析を網羅しています。さらにOlink Visualizerで統計解析データを簡単に可視化できる点が大きな特徴です。
この機会に理研ジェネシスのOlinkプロテオーム解析を試してみませんか。
Olinkプロテオーム解析についてのお問い合わせはこちらから → お問い合わせフォーム
最後に
弊社では、上記の統計解析に加え、対応ありのt検定、相関分析、二次元配置分散分析にも対応しています。これらの解析を駆使することで、より深いデータ解釈と新たな発見につながる可能性を広げます。詳細な内容は次回以降のブログでご紹介していきます。
2025年も変わらぬご支援をどうぞよろしくお願いします。

