特長
- Single Nucleotide Variation(SNV)、Short Insertion/Deletion(InDel)の探索と弊社が独自開発したシステムによるアノテーション付与。
- 遺伝性疾患からがん※まで候補遺伝子の同定に有用。
- 腫瘍検体用のオプション解析として、マッチドペア解析、TMB算出をご提供可能。
- ヒト腫瘍細胞をマウスに移植したXenograftサンプルの変異解析も実施可能。
解析概要
ご提供いただくHWESのFASTQファイルを参照配列にマッピングした後、SNVを検出し、アノテーションの付与を行います。複数サンプルの場合はサンプル間比較用のリストを作成することも可能です。体細胞変異検出を目的としたマッチドペア解析やヒト腫瘍細胞をマウスに移植したXenograftサンプルの変異解析も実施可能です。
解析結果
ご提供いただくHWGSのFASTQファイルを参照配列に対してマッピング
※1し、変異箇所(SNV
※1)を検出します。 検出されたSNVは、各種アノテーション
※2をつけてエクセル・マクロファイル
※3 とテキストファイルでご提出します。
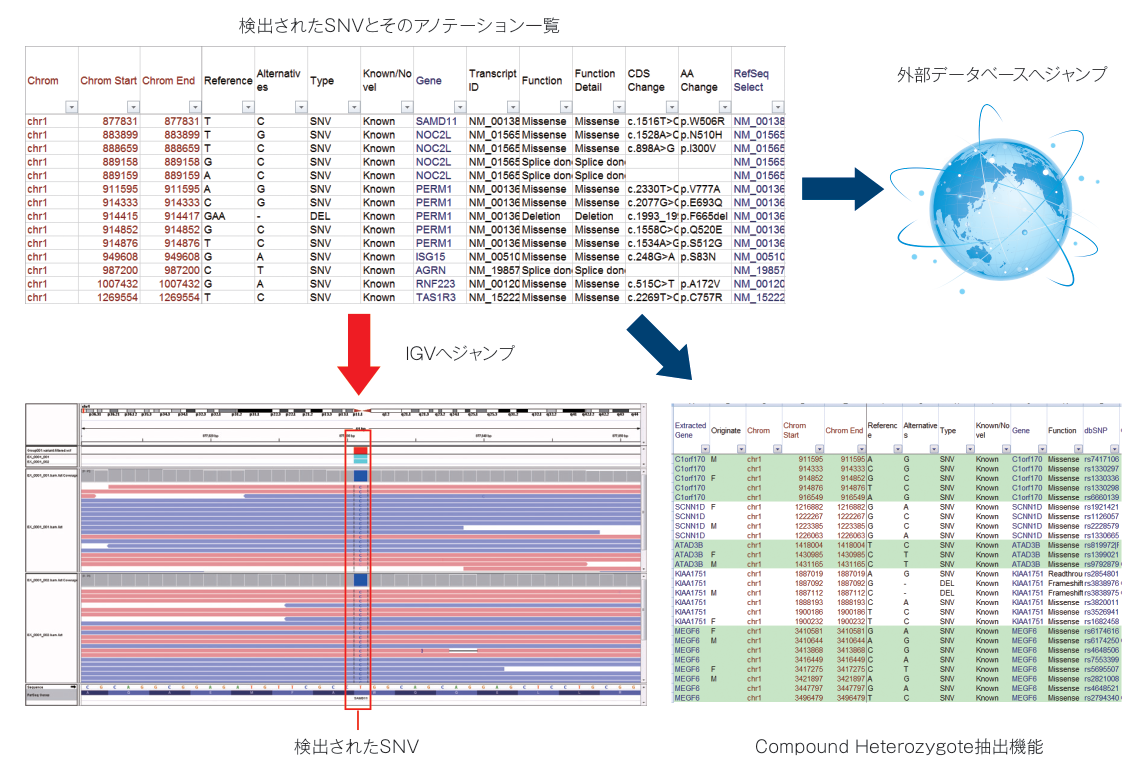
- ※1 マッピング結果、SNV、DepthはIntegrative Genomics Viewer(IGV)で閲覧することが可能です。
- ※2 検出されたSNVには次のようなアノテーションがつきます。 変異箇所、塩基、新規/既知、影響を与える遺伝子、遺伝子への影響、dbSNP ID、RefSeq ID、CCDS ID、アレル頻度(gnomAD、1000 Genomes、HGVD、GEM-J WGA)、Conserved Elements、アミノ酸置換影響予測(SIFTなど)、Splice Site変異影響予測、Depth、クオリティ値、サンプルごとのジェノタイプデータなど。
- ※3 アノテーションは弊社オリジナルのエクセル・マクロ(AnnotationViewer)で観覧でき、IGVおよび外部データベースと連携するようになっています。
解析条件
イルミナ社シーケンサーより出力されたpaired-endのFASTQファイル(生殖細胞変異の解析では平均depth 100x以上、体細胞変異の解析では平均depth 200x以上を推奨)
納品物
- 解析報告書
- データHDD:リード情報(FASTQ)、マッピングデータ(BAM)、SNV/InDel/SV/CNV検出データ(VCF)、SNV/InDel/SV/CNVのアノテーションデータ(エクセル・マクロファイルとテキストファイル)

