ネオアンチゲン探索
特長
- ネオアンチゲン探索では、がん細胞特異的な遺伝子変異情報とRNA発現量情報を組み合わせた解析により、ネオアンチゲンのペプチド配列の予測を行います。
- ネオアンチゲン※を標的としたがん免疫治療法の開発、研究にご活用いただけます。
- 本解析は、協業先(株式会社スタージェン)で実施します。

- ※ がん細胞は変異由来の新たな抗原(ネオアンチゲン)を細胞表面に提示しています。ネオアンチゲンは中枢性免疫寛容を免れているため、強い免疫応答の誘導が期待できます。そのため、現在、ネオアンチゲンを標的としたがん免疫治療の開発が進められています。
解析概要
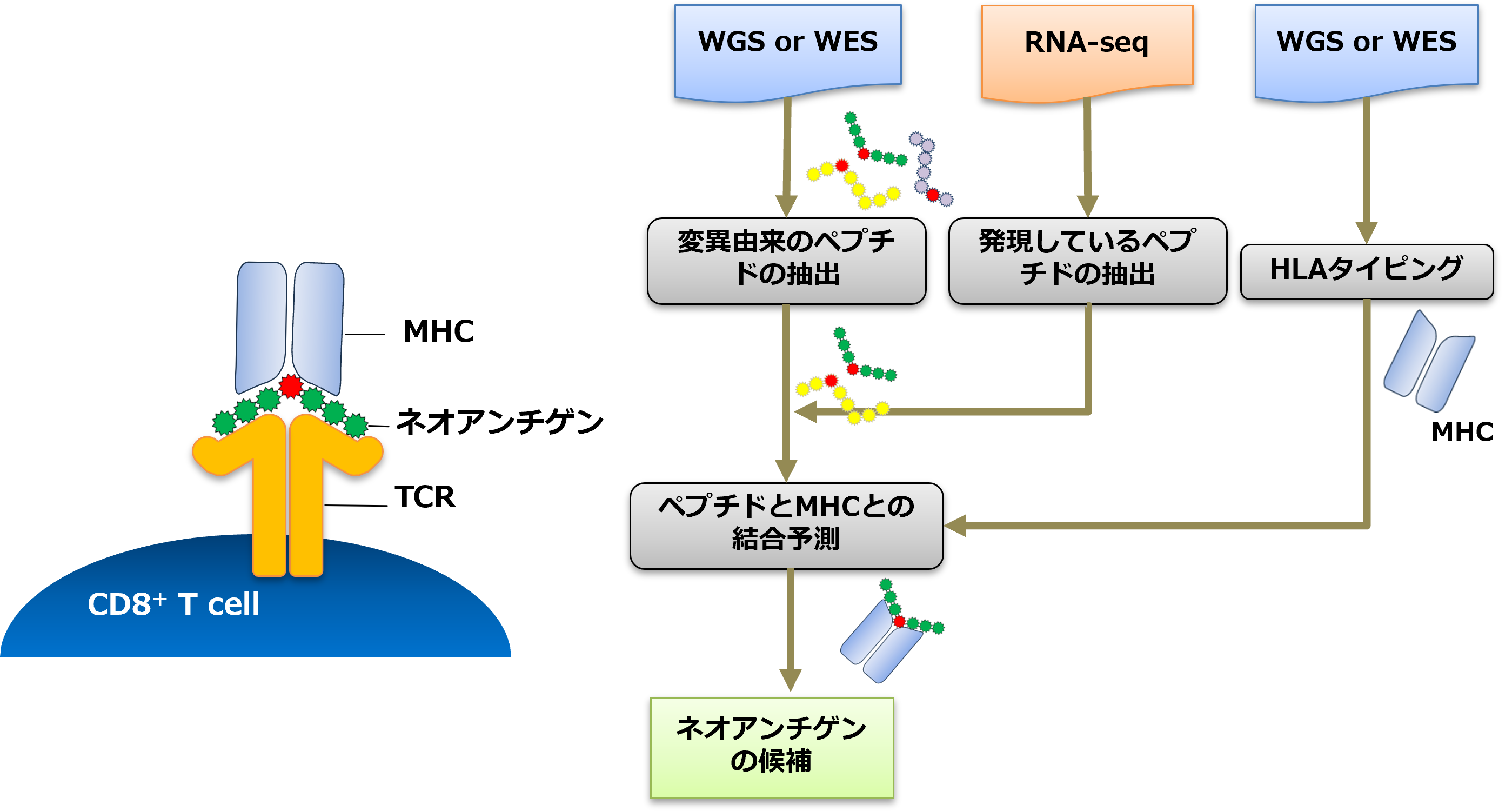
図1. ネオアンチゲン探索の解析フロー
解析に使用するデータの種類によって、以下の2種類の解析パターンをご用意しております。ご希望の解析パターンをご選択ください。
(1)ゲノム解析+RNA-seq解析(融合遺伝子、発現解析)
ゲノムデータとRNA-seqデータから、遺伝子変異情報とRNA発現量情報を組み合わせて、ネオアンチゲンの探索を行います。
※ HLAタイピングデータが必要です。お手元にデータがない場合は、弊社でHLAタイピングを実施します。
(2)ゲノム解析(RNA-seqのデータが得られない場合)
ゲノム情報のみを使用してネオアンチゲンの探索を行います。
※ HLAタイピングデータが必要です。お手元にデータがない場合は、弊社でHLAタイピングを実施します。
*HLAタイピング(オプション)
ゲノムデータ(BAMファイル)からHLAタイピングを行います。
詳細はこちら
解析結果・納品物
(1)ゲノム解析+RNA-seq解析(融合遺伝子、発現解析)
- 検出された変異周辺から翻訳されるアミノ酸配列の情報(テキストファイル)
- 各ペプチドとMHCクラスI分子との結合予測スコアの情報(テキストファイル)
- 特徴的なmutantペプチドのパターンの出現頻度の情報(テキストファイル)
- アミノ酸配列のFASTAファイル、既知エピトープとのホモロジー探索結果(テキストファイル)、T細胞受容体との結合・相互作用予測結果(テキストファイル)
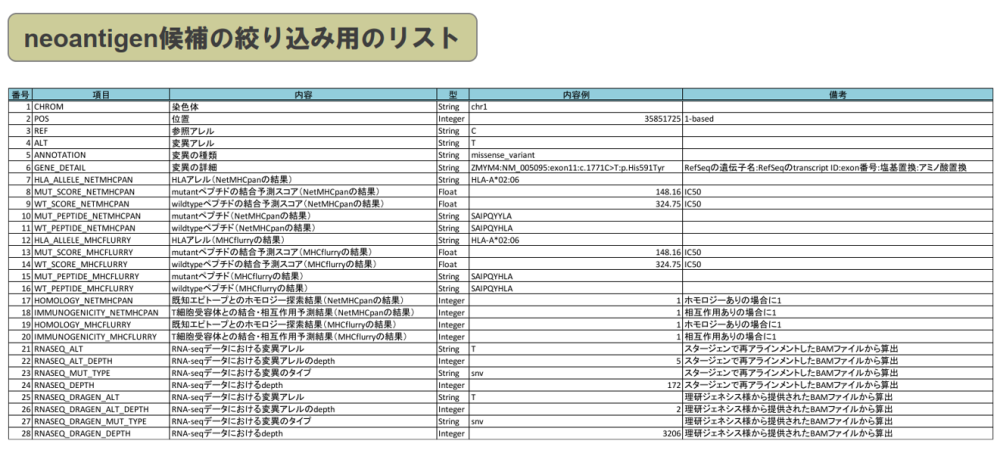
- RNA-seqデータ解析の結果を使用したneoantigen候補の絞り込みリスト(テキストファイル)
(2)ゲノムデータ解析(RNA-seqのデータが得られない場合)
- 検出された変異周辺から翻訳されるアミノ酸配列の情報(テキストファイル)
- 各ペプチドとMHCクラスI分子との結合予測スコアの情報(テキストファイル)
- 特徴的なmutantペプチドのパターンの出現頻度の情報(テキストファイル)
- アミノ酸配列のFASTAファイル、既知エピトープとのホモロジー探索結果(テキストファイル)、T細胞受容体との結合・相互作用予測結果(テキストファイル)
*HLAタイピング(オプション)
- 各検体のHLA各座位のタイピング結果(テキストファイル)。
解析条件
- Human Whole Genome Sequencing(HWGS、全ゲノム解析)
詳細はこちら - Human Whole Exome Sequencing(HWES、がんエクソーム解析)
詳細はこちら - RNA sequencing (RNS-seq)
詳細はこちら - HLAタイピング
詳細はこちら
※ HWGSまたはHWESのどちらかをご選択ください。
(2)本解析のみをご利用の場合
- 腫瘍検体由来のVCFファイル
- 遺伝子発現データ(DRAGENから出力した.quant.sfファイル)
- 融合遺伝子データ(STAR-Fusionから出力したTSVファイル)
- 遺伝子発現解析および融合遺伝子解析で使用したBAMファイル
- 正常検体由来のHLAタイピングデータ(テキストファイルやExcelファイルなどの形式で,第2区域までの遺伝型の情報を含む)
解析例